2 Matériel et Méthodes
2.1 Identification de la maladie
Pour la mise au point de la méthode, les feuilles analysées doivent avoir un statut connu vis-à-vis de la maladie. Pour ce faire, la méthode de détection par PCR (Polymerase Chain Reaction) est pour l’instant la plus utilisée pour surveiller l’évolution de la maladie. Cette méthode permet de détecter l’ADN de la bactérie à partir d’échantillons de feuilles (Gottwald, 2010). L’inconvénient de cette méthode est qu’elle ne permet pas le traitement d’un volume important d’échantillons et ne donne pas une réponse immédiate sur le prélèvement effectué sur le terrain. La PCR est une technique reconnue de multiplication de l’ADN d’un échantillon. Le but est de dupliquer les échantillons d’ADN prélevés à chaque cycle de réaction via l’ADN polymérase. Ainsi, avec un seul brin d’ADN de départ, des millions de répliques sont produites à chaque cycle de réplication. La qPCR repose sur le même principe avec des améliorations de la technique de départ. Le principe est toujours de multiplier l’ADN de l’échantillon, mais avec cette fois une quantification du nombre de cycles PCR nécessaires à la détection de la maladie. Le nombre de cycles de réplication ou “Cycle Threshold” (CT) correspond au nombre de cycles PCR à partir duquel le produit de la PCR est détectable. Ainsi, plus le CT est faible plus il y a d’ADN présent initialement et inversement. Les échantillons sains et malades peuvent donc être distingués selon leur valeur de CT selon un seuil fixé. La méthode officielle actuelle basée sur la PCR classique est actuellement en cours de remplacement par une méthode basée sur la qPCR et qui utilisera le seuil de 36 cycles de détection maximum pour identifier un échantillon comme étant porteur de la maladie. C’est ce seuil de 36 cycles ou CT36 qui sera utilisé dans cette étude pour statuer de l’état malade ou sain des arbres. Chaque échantillon est testé 3 fois (on parle de triplicats techniques) et chaque plaque de 96 puits de qPCR contient trois témoins négatifs et deux témoins positifs. Lorsque les témoins donnent des résultats inattendus ou que les triplicats ne sont pas cohérents, les échantillons sont repassés.
2.2 Choix des arbres
Le choix des parcelles échantillonnées a été conditionné par la recherche d’un équilibre entre les arbres malades et sains sur chaque parcelle basée sur les données de la surveillance officielle et du Cirad. Le défi est que les statuts des arbres ne sont pas connus à l’avance et il peut être compliqué de faire un design équilibré en amont de la récolte de données. L’objectif a donc été dans un premier temps de trouver les agriculteurs qui possèdent les trois variétés d’agrumes sur lesquelles se base cette étude, à savoir les citrons (Citrus limon), les mandarines zanzibar (Citrus reticulata) et les tangors (Citrus reticulata x sinensis). Dans un deuxième temps, pour chacun de ces agriculteurs il a fallu identifier précisément le statut des arbres.
Concernant les caractéristiques altitudinales et pluviométriques des parcelles, celle de Mr Hoarau et Mr Pothin échantillonnées à Petite-Ile sont comprises dans une échelle de pluviométrie identique la parcelle de Mr Gonthier échantillonnée dans les hauts du Tampon à savoir entre 1430 et 1680 mm de pluie par an (Equipe Artists - UR AïDA, 2021). Leurs altitudes respectives sont cependant très différentes avec une différence de 1000 m et plus entre la parcelle du Tampon et les autres (figure 2.1). La parcelle de Mr Barret située au niveau de la mer est la parcelle qui reçoit le moins de précipitations avec seulement 930 à 960 mm de pluie par an (Equipe Artists - UR AïDA, 2021).
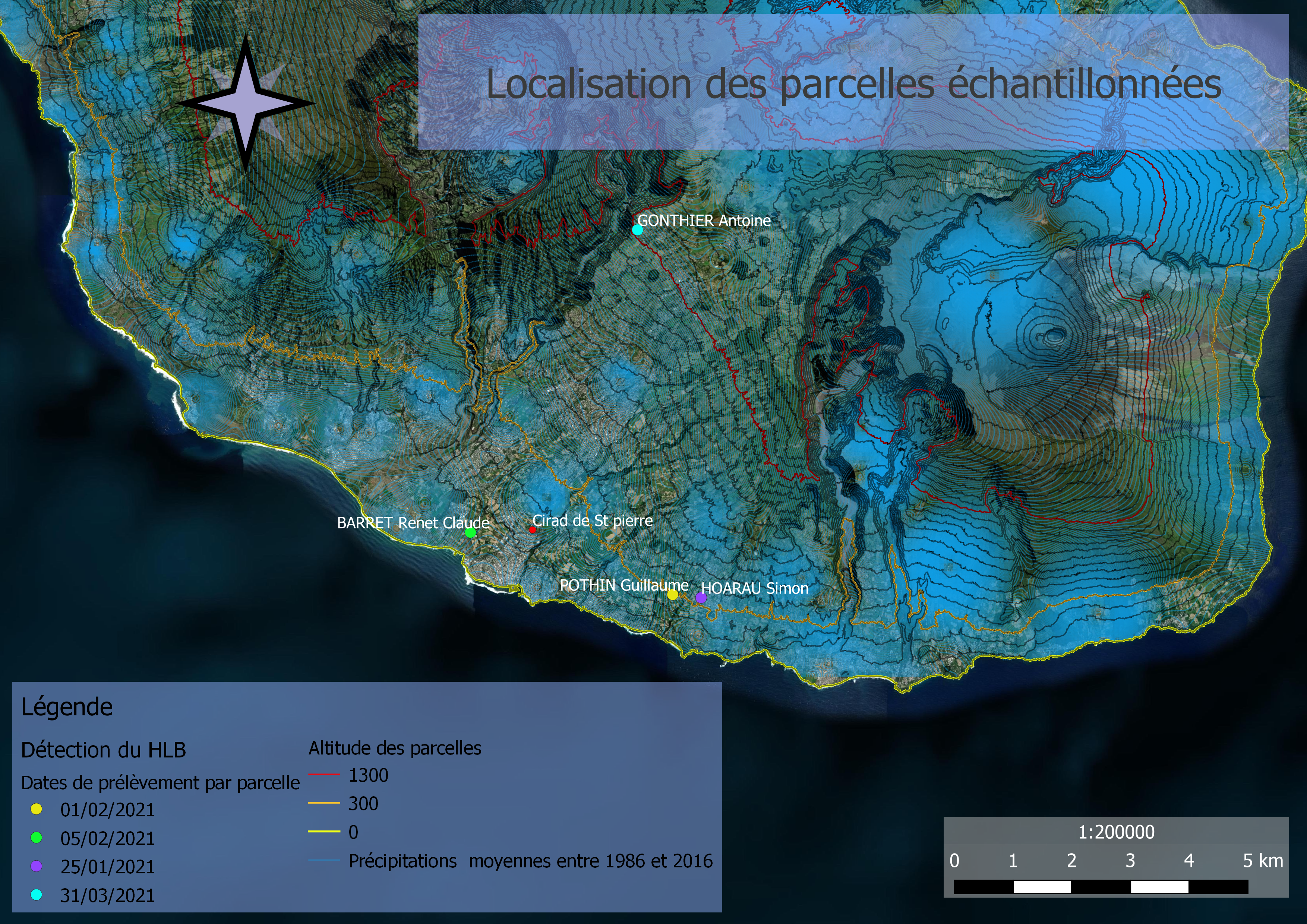
Figure 2.1: Carte présentant les parcelles échantillonnées, leurs dates de prélèvement ainsi que la pluviométrie et les altitudes
L’échantillonnage a commencé dans la commune de Petite-Île, qui est la plus grosse zone de production d’agrumes de l’île (environ 25% de la surface en agrumes) (Guilloteau, 2018). Cette zone est aussi très touchée par le HLB (près de deux parcelles sur trois) avec par conséquent, des prospections importantes de la part de la DAAF et de la FDGDON qui fournissent des données précieuses pour l’échantillonnage. Les parcelles de Mr Hoarau et Mr Pothin échantillonnées à Petite-Île possèdent les trois variétés recherchées mais ont une répartition différente entre les arbres malades et sains. Dans la parcelle de Mr Hoarau l’échantillonnage s’est basé sur une première cartographie des statuts HLB des arbres réalisée dans le cadre d‘une expérimentation de la FGDGON pour le développement d’une autre méthode moléculaire. 42 arbres ont ainsi été sélectionnés dont 19 arbres se sont révélés sains et 23 arbres se sont révélés infectés par le HLB (figure 2.2).
Figure 2.2: Échantillonnage réalisé dans la parcelle de Mr Hoarau en 2021
A l’inverse dans la parcelle de Mr Pothin, l’échantillonnage des arbres s’est basé sur les dires d’expert de l’agriculteur et trois arbres sains pour 39 arbres infectés par le HLB ont été trouvés (figure 2.3).
Figure 2.3: Échantillonnage réalisé dans la parcelle de Mr Pothin en 2021
La parcelle de Mr Barret située à Saint Pierre et constituée uniquement de citronniers a été échantillonnée de manière exhaustive. Cette parcelle fut la plus analysée en laboratoire. Les 156 arbres de la parcelle ont d’abord été regroupés par pools de 5 puis certains arbres ont été analysés individuellement. Au final 10 citronniers sains et quatre infectés par le HLB ont été trouvés (figure 2.4).
Figure 2.4: Échantillonnage réalisé dans la parcelle de Mr Barret en 2021
A l’issue de cet échantillonnage, un grand nombre d’arbres infectés chez Mr Pothin et Barret ont été trouvés, il y avait donc un déficit d’arbres sains. La parcelle de Mr Gonthier située au Tampon a donc été trouvée pour pallier à ce déficit. Cette parcelle compte une répartition égale entre les trois variétés et dont des lots d’arbres ont été testés négatifs aux HLB en novembre 2020 lors de la surveillance officielle. Pour chaque variété 14 arbres ont été échantillonnés (figure 2.5) afin de rééquilibrer l’ensemble du jeu de données. Par manque de temps, ces arbres n’ont pas été re-testés en qPCR et ont été considérés comme négatifs. Cet échantillonnage n’est pas idéal, dans la mesure où les facteurs “agriculteur” et “variété” ne sont pas croisés, et une attention particulière sur ce point devra être portée dans l’interprétation des résultats.
Figure 2.5: Échantillonnage réalisé dans la parcelle de Mr Gonthier en 2021
Au final, au cours de cette étude 140 arbres ont été échantillonnés et numérotés de A1 à T7.
2.3 Spectroscopie à main
L’objectif de l’utilisation de la spectroscopie est la discrimination des arbres sains et malades de façon rapide et clairement identifiable en passant l’outil de détection sur la parcelle. La détection de la maladie sur les plantes repose sur la capacité des feuilles à absorber, réfléchir ou transmettre la lumière dans différentes longueurs d’onde mesurées en nanomètre (nm). Ces caractéristiques sont liées à la composition biochimique des feuilles. Les variations de mesure de spectre peuvent donc être expliquées dans les différents domaines du spectre par la teneur en pigment des feuilles dans le visible (400-750 nm), par la structure cellulaire avec l’essentiel des vaisseaux dont le phloème dans le proche infrarouge (750-1250 nm) et par la teneur en eau dans le “Short-wave infrared” ou SWIR (1250-2500 nm) comme indiqué sur la (figure 2.6 (De Blomac, 2014)) (Comar, 2013). Les bandes spectrales du visible (bleu, vert, rouge, “red-edge”) sont les plus utilisées dans la détection de maladie des feuilles (Mishra et al., 2007). La qualité des résultats dépend de la résolution du capteur utilisé. Dans le visible (400-750 nm), la coloration des feuilles est due à la teneur en chlorophylles, caroténoïdes et anthocyanes. Les chlorophylles (de type a et b) captent la lumière dans des longueurs d’ondes correspondant au bleu et au rouge. Les caroténoïdes captent la lumière exclusivement dans le bleu et les anthocyanes la captent exclusivement dans le vert. Vers la fin du domaine visible (700 nm) il n’y a plus de captage de lumière aussi important et la lumière est quasiment entièrement réfléchie dans cette zone appelée “red edge”. Ensuite, la zone du proche infrarouge (750-1250 nm) correspond principalement à la structure cellulaire interne des feuilles qui est aussi peu affectée en cas de maladie. Cette zone est utile pour la discrimination des feuilles entre feuillus et résineux qui ont des structures de tissus différentes. Enfin, dans le SWIR (1250-2500 nm), c’est principalement la teneur en eau des feuilles qui est mesurée. Plus la réflectance est faible, plus la teneur en eau est grande, ce qui est utile pour caractériser un stress hydrique chez la plante (Albetis de la Cruz, 2018).
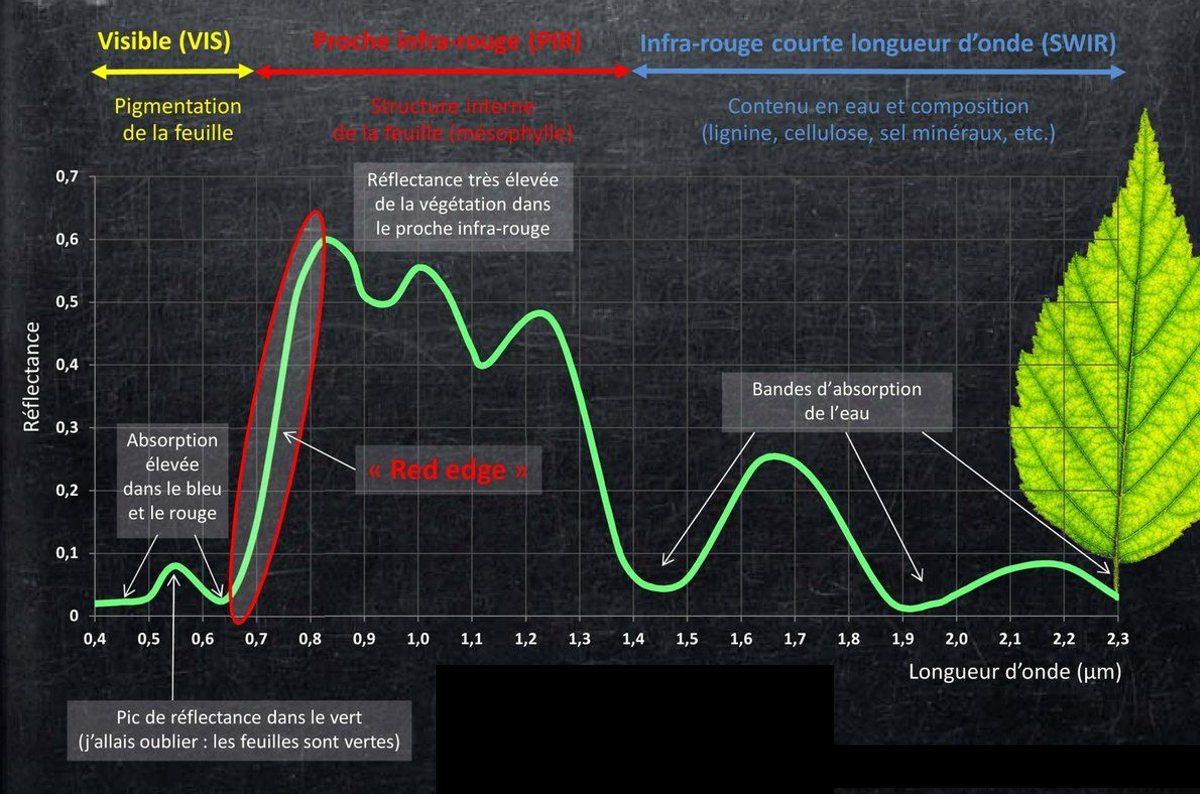
Figure 2.6: Schéma de la variation de la réflectance de la végétation
Au cours de cette étude, la détection du statut infecté en HLB des arbres va être réalisé par la méthode de la Spectroscopie Proche Infra Rouge (SPIR) à main qui est catégorisée comme étant de la proxidétection (Albetis de la Cruz, 2018). Dans le cas de la proxidétection, l’acquisition des données se fait par contact direct ou à quelques centimètres de l’objet cible à l’échelle de la feuille ou de la plante (figure 2.7).
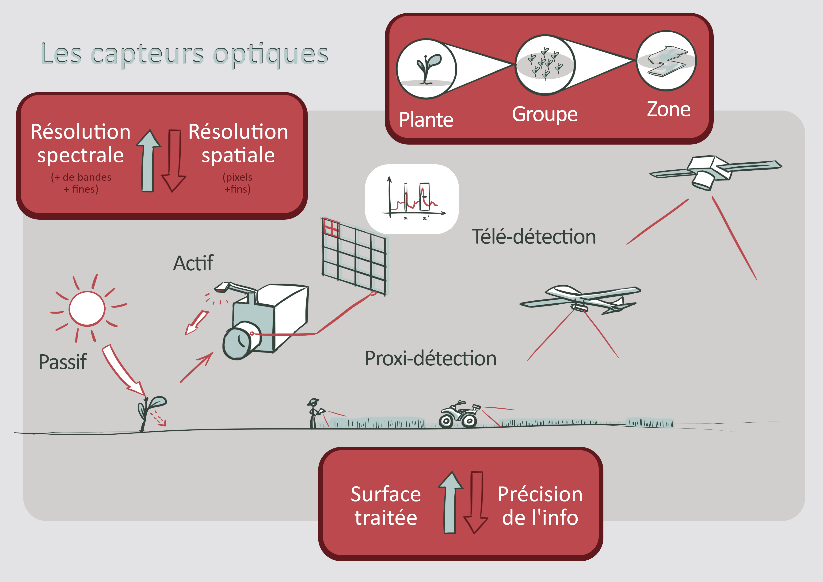
Figure 2.7: Schéma d’application des capteurs optiques (Source personnelle)
Ainsi, pour l’acquisition des données, un spectromètre de terrain (ASD fieldspec Pro) a été utilisé en laboratoire (figure 2.8). L’appareil est utilisé afin de mesurer la réflectance, une mesure correspondant à la proportion (comprise entre 0 et 1) de lumière réfléchie par la surface d’un matériau qui est ici une feuille d’agrume (Comar, 2013). Afin de mesurer cette réflectance, la feuille disposée sur un disque blanc en Spectralon® qui renvoie quasiment toute la lumière transmise. Cette lumière transmise par l’appareil traverse d’abord la feuille puis est réfléchie et retraverse la feuille avant d’atteindre le capteur. Le Spectralon® est régulièrement nettoyé au papier de verre pour conserver sa valeur de réflectance constante au cours du temps. De plus, les mesures sont réalisées à température ambiante et un blanc est effectué toutes les 15 minutes en réalisant une mesure de SPIR sur le support en Spectralon® seul. L’appareil mesure une bande de longueur d’onde allant de 350 à 2500 nm, la courbe caractérisant la réflectance en fonction des longueurs d’onde mesurées est appelée spectre de réflectance (Comar, 2013). Pour chaque mesure, le spectre de réflectance correspond à une moyenne de 50 mesures de réflectance effectuées par l’appareil entre 350 et 2500 nm.

Figure 2.8: Photographie d’un spectromètre proche infrarouge à main
Sur chaque arbre 10 feuilles sont prélevées de manière aléatoire autour de l’arbre. Le nombre de 10 feuilles à échantillonner provient d’un compromis entre le temps d’analyse de ces feuilles et de la précision des résultats recherchés. Ce compromis n’aurait pas pu être possible sans les travaux d’un précédent stagiaire, Nathan Créquy qui avait prélevé trois lots de 10 feuilles sur 14 arbres échantillonnés et sur lesquelles des tests sur la baisse de précision ont pu être effectués avant d’aboutir au nombre idéal de feuilles à prélever (Créquy, 2020).Sur chacune des 10 feuilles prélevées par arbre, six mesures de réflectance sont réalisées afin d’avoir des spectres de réflectance issus des différentes zones de la feuille. Ce nombre de mesures optimal a été calculé en amont via les données issues de l’étude de Nathan Créquy qui avait 10 mesures SPIR par feuille et dont les paramètres de précision furent comparés au moyen de tests statistiques pour chaque mesure effectuée par feuille (Créquy, 2020).En fonction des longueurs d’onde étudiées, les capteurs utilisés sont différents et regroupés en deux catégories, les capteurs multispectraux et hyperspectraux. Les capteurs multispectraux permettent d’enregistrer des bandes spectrales qui ne sont pas contiguës et qui peuvent être ciblées, par exemple : le proche infrarouge, le bleu, le rouge et le vert. A l’inverse, les capteurs hyperspectraux enregistrent plus ou moins toutes les bandes spectrales (en fonction de la résolution de l’appareil) de façon contiguë dans un intervalle de longueur d’onde compris entre 350 et 2500 nm (Bertaux, 2015). Au cours de cette étude, seul le capteur hyperspectral sera utilisé sur la partie SPIR avec six mesures SPIR par feuille sur les 10 feuilles prélevées par arbre. Le jeu de données (annexe 1) est donc composé des 140 arbres échantillonnés soit 1400 feuilles échantillonnées et analysées en SPIR. Cependant, le nombre réel de données comprenant les 6 répétitions SPIR par feuilles échantillonnées est de 8400.
2.4 Traitement des données par analyses statistiques et apprentissage supervisé
Tous les traitements statistiques sont réalisés sous R (R Core Team, 2021).
Les données utilisées pour les analyses sont les valeurs de réflectance issues de la SPIR à main allant de 350 à 2500 nm.
Dans un premier temps, l’objectif va être de caractériser les sources de variations des spectres de réflectance, et en particulier, l’influence des parcelles et des variétés sur ces derniers. Ainsi pour chaque longueur d’onde, une décomposition de la variance est mise en œuvre via une ANOVA à un facteur où la variable expliquée est la réflectance observée pour cette longueur d’onde et la variable explicative est la variété ou la parcelle. Cette analyse produit pour chaque longueur d’onde une valeur du F de Fisher correspondant au rapport entre la variance intermodalités (entre parcelles ou entre variétés) et la variance résiduelle (au sein des parcelles ou des variétés). Plus le F de Fisher est élevé, plus la variable testée (parcelle ou variété) affecte la réflectance. Dans un second temps, une ANOVA à trois facteurs (statut HLB, variété, parcelle) est réalisée afin de montrer l’influence du statut HLB en interaction avec la variété et la parcelle sur le spectre de réflectance global.
Une fois démontrée l’influence ou la non-influence de ces variables de nuisance et l’effet qu’a le statut HLB sur le spectre de réflectance, il est possible d’envisager une prédiction sur le statut HLB des arbres à partir des valeurs de réflectance. Pour ce faire, trois types de méthodes de classification par apprentissage supervisé ont été mises en œuvre : les forêts aléatoires (RF pour « Random Forest» ), la machine à vecteurs de support (SVM pour « Support Vector Machine ») et la régression par les moindres carrés partiels (PLS pour « Partial Least Square »).
Un algorithme d’apprentissage supervisé se compose à la fois de données d’entrée “training set” (75% de la base d’apprentissage) et de données cibles “test set” (25% de la base d’apprentissage) (Tuszynski, 2020). Les données d’entrée comprennent des spectres de réflectances associés à un statut HLB. Elles permettent à l’algorithme de « s’entrainer » à reconnaître le statut HLB des arbres à partir des spectres de réflectance. Les données cibles sont composées de spectres pour lesquels le statut HLB est connu mais ne peut être utilisé par l’algorithme car ces données sont utilisées pour la vérification et pour éviter la redondance d’utilisation d’une donnée dans l’apprentissage. Elles permettent de mesurer la performance de l’algorithme. Après s’être assuré de la bonne performance de l’algorithme, on peut l’utiliser pour prédire le statut HLB d’arbres dont on ne dispose que du spectre de réflectance (Knaus, 2015).
2.5 Forêts Aléatoires (RF)
Les RF est une technique utilisant un algorithme d’apprentissage supervisé couplé avec un arbre de décision (Liaw et Wiener, 2002) (Borkovec et Madin, 2019). Un arbre de décision est une construction de choix souvent binaire qui permet de prendre une décision. Cet algorithme permet de créer une forêt d’arbres de décision qui permet d’améliorer la généralisation (sur un paramètre recherché) de l’ensemble du modèle. La RF combine la simplicité de lecture des arbres de décision ainsi que la robustesse de l’apprentissage supervisé qui améliore la précision de la prédiction (Moutarde, 2017). La première étape de la construction du RF est la création d’un jeu de données pour chaque arbre de décision (figure 2.9 (Moutarde, 2017)). Ce jeu de données se compose des lignes des données originelles piochées aléatoirement (la même ligne pouvant être sélectionnée plusieurs fois comme la ligne 2 de l’exemple en rouge). Ce procédé augmente la variété des arbres de décision ce qui rend l’algorithme encore plus robuste (Moutarde, 2017).
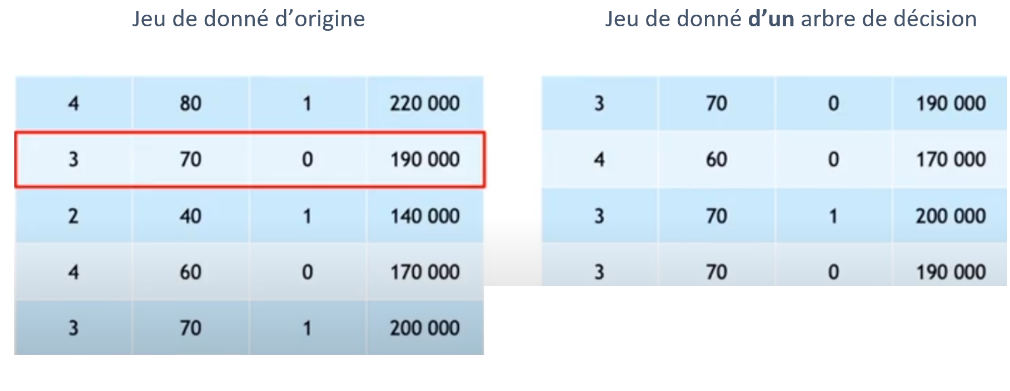
Figure 2.9: Schéma 1 de construction d’un jeu de données d’un arbre de décision en RF
La deuxième étape va être la création de chaque arbre de décision sur une partie différente (aléatoire et non exclusive) des données et des variables à l’intérieur de leurs jeux de données (figure 2.10, rectangles de couleurs (Moutarde, 2017)). L’intérêt est d’avoir des estimateurs différents dans chaque arbre de décision pour avoir un cheminement différent (Moutarde, 2017).
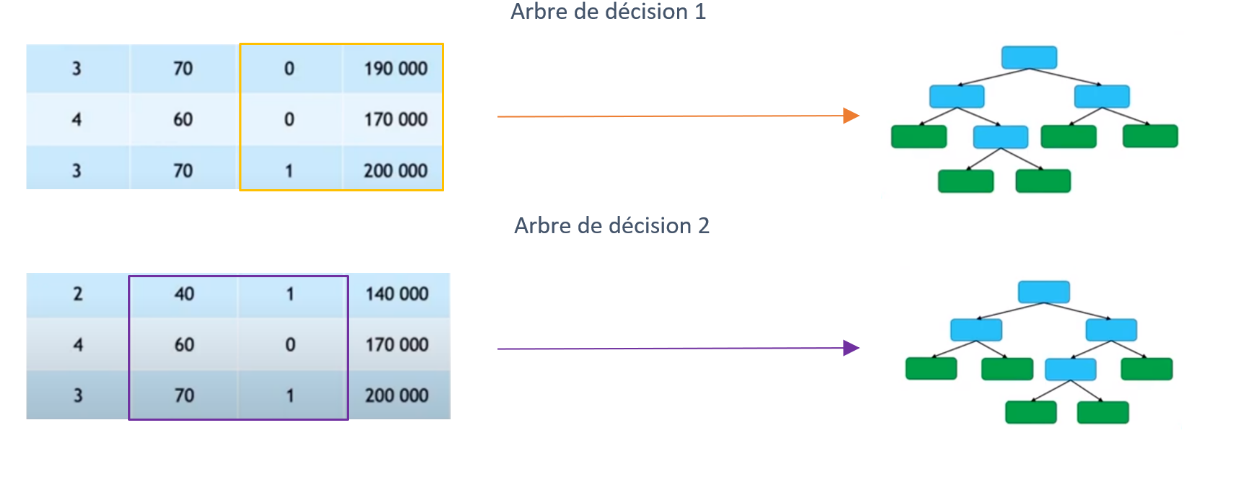
Figure 2.10: Schéma 2 de construction d’un arbre de décision en RF
La troisième étape consiste à répéter l’étape 1 et 2 afin de créer autant d’arbres de décision que l’on souhaite dans le but d’obtenir un ensemble (forêt) d’arbres de décision générés aléatoirement (figure 2.11 (Moutarde, 2017)). Le résultat de la RF va correspondre à la moyenne des décisions de l’ensemble des arbres pour l’élément recherché.
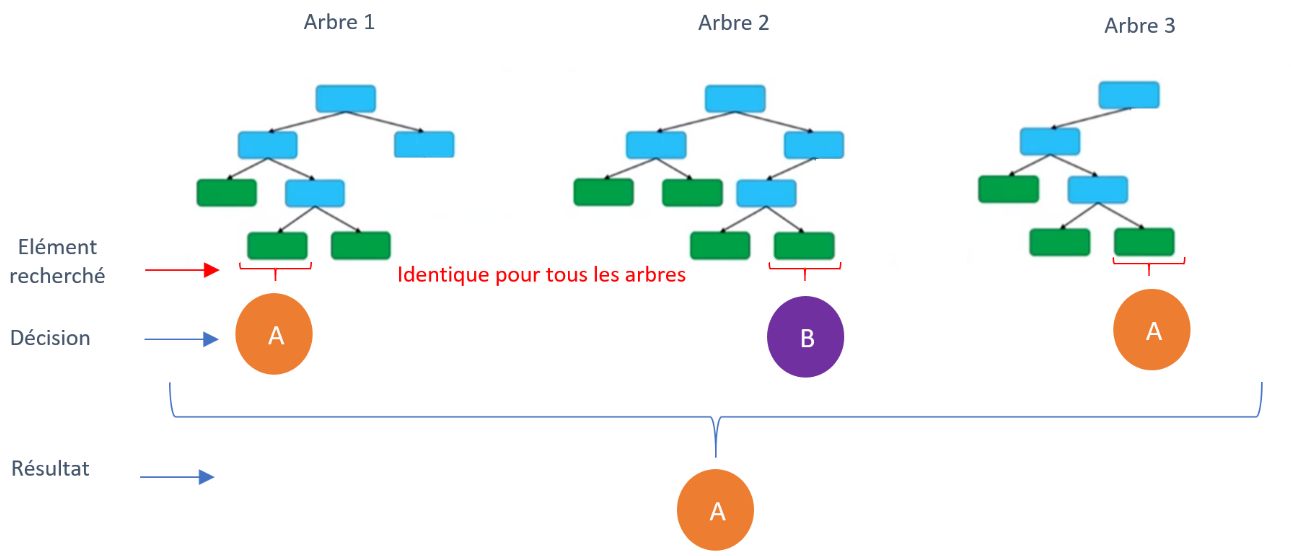
Figure 2.11: Schéma de construction d’une Forêts Aléatoire
2.6 Machine à Vecteurs de Support (SVM)
La SVM est aussi un algorithme d’apprentissage supervisé qui permet de faire des prédictions sur des variables qualitatives ou quantitatives (Meyer et al., 2020). L’objectif du SVM va consister à séparer deux classes de données, ici (infecté ; sain) pour pouvoir ensuite établir une généralisation dans la prédiction quand on ne connaîtra pas la nature de l’échantillon (infecté ; sain). Pour séparer ces deux classes, l’idée est de maximiser les distances entre les échantillons avec un hyperplan séparateur (appelé aussi « support vector ») (figure 2.12 (Cortes et Vapnik, 1995)).
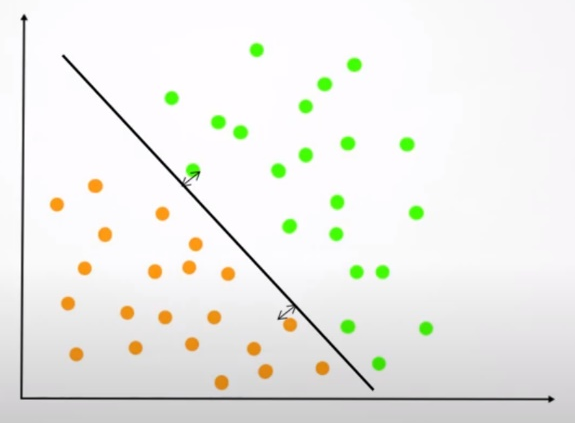
Figure 2.12: Schéma de construction d’un hyperplan dans le cadre du SVM
L’apprentissage supervisé va permettre de déterminer l’équation de l’hyperplan qui séparera les jeux de données des individus sains de ceux des malades. Cela va ensuite permettre la prédiction pour les jeux de données à tester.
2.7 Régression par les Moindres Carrés Partiels (PLS)
Pour finir la PLS est une méthode statistique permettant de traiter une variable binaire à expliquer (ici le statut HLB infecté ou sain) à partir de variables explicatives nombreuses (ici les mesures de réflectance pour chaque longueur d’onde) (Mevik et al., 2020).
L’idée derrière la régression PLS est de créer, à partir d’un tableau avec n observations (les statuts HLB) décrites par X variables explicatives (réflectances pour chaque longueur d’onde), un ensemble de h composantes correspondant aux fonctions de chaque réflectance (Tenehaus, 1999).
Cette méthode consiste à rechercher dans un premier temps des composantes h, expliquant au mieux à la fois les variables explicatives X et les variables à expliquer Y. Les équations de régression PLS sont ensuite obtenues en régressant chaque variable Y sur les composantes h, puis en exprimant ces régressions en fonction des variables X d’origine (figure 2.13 (Tenehaus, 1999)).
Figure 2.13: Tableau représentant les coefficients de régression en fonction de X et Y
L’apprentissage supervisé va permettre de déterminer les équations de régression PLS nécessaires à prédire les jeux de données à tester.
2.8 Performance des classifications
En apprentissage supervisé la matrice de confusion mesure la qualité d’un système de prédiction (table 2.1).
| Négatif confirmé | Positif confirmé | |
|---|---|---|
| Négatif prédit | Vrai Négatif (VN) | Faux Négatif (FN) (Erreur de type 2) |
| Positif prédit | Faux positif (FP) (Erreur de type 1) | Vrai positif (VP) |
De cette matrice, plusieurs paramètres de performance sont calculés dont :
La qualité de la prédiction (“Accuracy”) = VP + VN / VP + VN + FN + FP met en évidence les erreurs de la prédiction qu’elles soient de type 1 ou de type 2
La précision (“Precision”) = VP / VP + FP met en évidence les erreurs de type 1
L’erreur de type 1 est problématique dans le cadre de la maladie, car elle implique l’arrachage d’un arbre sain alors prédit comme étant positif au HLB
- La sensibilité (“Sensitivity”) = VP / VP + FN met en évidence les erreurs de type 2
L’erreur de type 2 impacte quant à elle la détection de la maladie, l’arbre est prédit comme étant négatif à la maladie alors qu’il est en réalité malade.
Ces paramètres permettent de rendre compte de l’efficacité de la prédiction et de pouvoir choisir la meilleure méthode de prédiction.
Ces méthodes ont été choisies à partir de la bibliographie où la qualité de la prédiction est par exemple de 85% pour SVM dans l’article de Sindhuja Sankaran (Sankaran et al., 2013) et 96.4% pour PLS dans l’article de Xiaoling Deng (Deng et al., 2020).
2.9 Amélioration du protocole de terrain
Afin d’améliorer le protocole de terrain pour de futures analyses, la question de savoir s’il était possible d’alléger le protocole de SPIR à main (i.e., faire moins de feuilles par arbre, moins de mesures de réflectance par feuille) sans perdre en performances de classification s’est posée. Pour ce faire, 100 jeux de données ont été simulés et dégradés par rééchantillonnage dans le vrai jeu de données et analysés avec les trois méthodes statistiques présentées.